Des millions d’images de plancton, trois communautés : comment la data science révèle les patterns cachés de l’océan
ACP, Clustering, Plancton, Analyses multivariées, Océanographie, Machine learning non supervisé, K-means, Typologie communautés
Des millions d’images de plancton, trois communautés : comment la data science révèle les patterns cachés de l’océan
De la collecte de données massives à la découverte de typologies globales : retour sur une analyse qui combine imagerie sous-marine et statistiques multivariées.
Cet article s’appuie sur mes travaux publiés dans Global Ecology and Biogeography :
Panaïotis, T., et al. (2023). Three major mesoplanktonic communities resolved by in situ imaging in the upper 500 m of the global ocean. Global Ecology and Biogeography, 32(11), 1961-1977. https://doi.org/10.1111/geb.13741
Des millions d’images, aucune structure apparente
L’océan couvre 70% de la surface de la Terre, mais on connaît encore mal la biodiversité qui l’habite. Le plancton, ces organismes qui dérivent au gré des courants, joue un rôle fondamental dans les réseaux trophiques marins et le cycle du carbone. Mais comment cartographier ces communautés à l’échelle mondiale ?
Entre 2008 et 2019, des équipes internationales ont collecté 6,8 millions d’images de plancton à travers tous les océans du globe avec l’Underwater Vision Profiler 5 (UVP5) (Picheral et al. 2010), un instrument d’imagerie sous-marine. Cette caméra prend des images en continu pendant sa descente dans l’eau, jusqu’à 6000 mètres de profondeur. Contrairement aux filets à plancton qui peuvent détruire les organismes fragiles, l’UVP5 les photographie sans les toucher.
Les données :
- 2 500 profils verticaux (Figure 1) à travers le globe dans les 500 premiers mètres de l’océan ;
- 330 000 organismes de plancton identifiés automatiquement grâce à des modèles de classification d’images par deep learning, entraînés et validés sur Ecotaxa (copépodes, Rhizaria, colonies de cyanobactéries…) ;
- pour chaque profil : abondances des différents groupes taxonomiques + variables environnementales (température, salinité…).
Les millions d’images collectées sont automatiquement classifiées par des modèles de deep learning (réseaux de neurones convolutifs). J’ai consacré un article scientifique entier à benchmarker différentes approches de classification, un sujet que j’aborde dans un autre article de blog !
Pendant mon stage de master 2, j’ai travaillé sur l’analyse de ces données. Comment en faire émerger des patterns cohérents ? Comment identifier des typologies de communautés qui aient du sens écologiquement ?
Trois grandes communautés planctoniques se cachaient dans les données, chacune associée à des conditions océanographiques bien spécifiques. Mais comment les révéler à partir de millions d’images ?
La stratégie : ACP + clustering
Une fois les données en main, j’ai appliqué un pipeline d’analyse classique en data science pour ce type de problème exploratoire.
1. Préparer les données
J’ai d’abord agrégé les images par profil vertical. Chaque profil devient une “observation” caractérisée par les abondances de chaque groupe taxonomique (copépodes, Rhizaria, Trichodesmium, appendiculaires, etc.).
Résultat : un tableau de contingence classique avec lignes = profils, colonnes = groupes taxonomiques.
Avant l’analyse, j’ai appliqué une transformation de Hellinger (Legendre et Legendre 2012) sur les abondances. C’est une étape souvent négligée mais essentielle quand on étudie des compositions de communautés. En effet, les abondances brutes posent problème : certains groupes sont naturellement très abondants (copépodes), d’autres rares (gros organismes). Sans transformation, l’ACP sera dominée par les groupes les plus abondants et on perdrait l’information sur la composition relative de la communauté.
La transformation de Hellinger convertit les abondances en proportions, puis applique une racine carrée. Ainsi, on analyse la composition de la communauté plutôt que les abondances brutes. Cette approche est particulièrement utile pour les analyses de communautés écologiques : elle préserve les distances écologiques tout en donnant du poids aux espèces rares.
2. Réduire la dimension (ACP)
J’avais 28 groupes de plancton en variables, souvent corrélées entre elles (certains groupes cooccurrent fréquemment). Difficile de visualiser et d’interpréter tout ça simultanément.
L’Analyse en Composantes Principales (ACP) condense cette information en quelques axes principaux qui capturent la majorité de la variance. C’est comme compresser une photo en JPEG : on garde l’essentiel de l’information en réduisant la taille.
J’ai conservé les 5 premières composantes principales (Figure 2), qui capturaient environ 65% de la variance totale. Au lieu de travailler avec 28 variables, je travaille avec 5 axes synthétiques qui résument les patterns principaux de composition des communautés.
Le choix de 5 composantes est pragmatique : les résultats de clustering restent similaires que j’en utilise 3, 5 ou 7.
3. Identifier les groupes (clustering)
Une fois les données réduites aux 5 premières composantes principales, j’ai appliqué un clustering hiérarchique avec la méthode de Ward pour regrouper les profils similaires.
La méthode de Ward minimise la variance au sein de chaque cluster, ce qui produit des groupes compacts et bien séparés. Le dendrogramme (arbre de classification) a révélé une structure claire : un grand saut entre 3 et 4 clusters, indiquant que 3 groupes (Figure 3) se distinguaient naturellement dans les données.
4. Valider et interpréter
La dernière étape est cruciale : les clusters ont-ils du sens écologiquement ?
J’ai validé le choix des 3 clusters avec :
- la cohérence du dendrogramme (grand saut = séparation nette) ;
- l’interprétation écologique (est-ce que ça fait sens ?).
Bonne nouvelle : les trois communautés identifiées correspondaient bien à des grands biomes océaniques distincts.
Trois communautés, trois océans
L’analyse a révélé trois grandes communautés planctoniques dans les 500 premiers mètres de l’océan mondial (Figure 4).
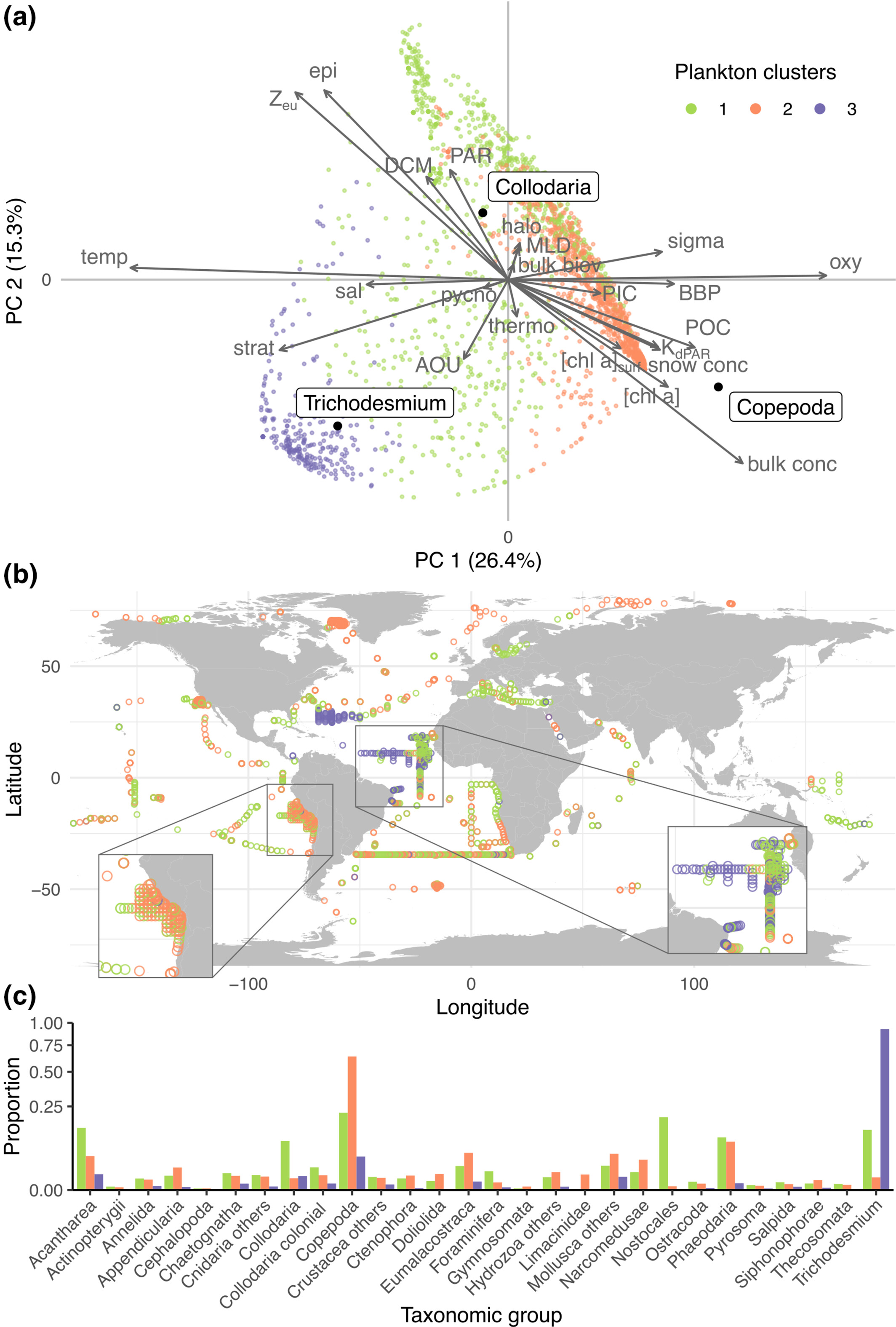
Communauté 1 : Enrichie en Trichodesmium
Localisation : Atlantique intertropical, eaux chaudes pauvres en nutriments
Organisme dominant : Colonies de cyanobactéries Trichodesmium, visibles à l’œil nu sous forme de filaments flottants
Rôle écologique : Ces cyanobactéries fixent l’azote atmosphérique et apportent cet élément vital dans des eaux autrement pauvres en nutriments. Elles forment ainsi la base des réseaux trophiques spécifiques dans les gyres subtropicaux.
Communauté 2 : Enrichie en copépodes
Localisation : Hautes latitudes (Arctique, Antarctique, Atlantique Nord) et zones d’upwelling (remontées d’eaux profondes riches en nutriments)
Organisme dominant : Copépodes, petits crustacés de quelques millimètres, les animaux les plus abondants de la planète
Rôle écologique : Les copépodes sont un maillon clé des réseaux trophiques marins : ils broutent le phytoplancton et sont consommés à leur tour par de plus gros organismes. Ces zones affichent une productivité biologique exceptionnelle.
Communauté 3 : Enrichie en Rhizaria
Localisation : Zones océaniques ouvertes pauvres en nutriments et certaines zones d’upwelling côtier
Organisme dominant : Rhizaria, protistes unicellulaires dotés d’un squelette minéral (silice ou carbonate de calcium)
Rôle écologique : Les Rhizaria sont des acteurs clés du cycle du carbone océanique. Leurs squelettes denses coulent rapidement, emportant le carbone organique vers les profondeurs. Ce mécanisme contribue au stockage à long terme du CO2 dans l’océan.
Ces trois communautés ne sont pas aléatoires : elles sont fortement corrélées aux conditions environnementales (température, nutriments…) et aux grandes régions biogéographiques de l’océan.
Le code en bref
Pour les curieux, voici le pipeline simplifié en quelques lignes. Le code complet avec toutes les vérifications et visualisations est disponible sur GitHub.
library(tidyverse)
library(vegan)
library(ggdendro)
# Transformation Hellinger + PCA
df_hel <- decostand(df_plankton, "hellinger")
pca <- rda(df_hel)
# Clustering hiérarchique (Ward) sur 5 premières composantes
scores_pca <- scores(pca, display = "sites", choices = 1:5, scaling = 1)
dist_euc <- dist(scores_pca, method = "euclidean")
clust <- hclust(dist_euc, method = "ward.D2")
clusters <- cutree(clust, k = 3)
# Composition taxonomique par cluster
composition <- bind_cols(df_plankton, cluster = clusters) %>%
pivot_longer(-cluster, names_to = "taxon", values_to = "conc") %>%
group_by(cluster, taxon) %>%
summarise(conc_mean = mean(conc), .groups = "drop") %>%
group_by(cluster) %>%
mutate(prop = conc_mean / sum(conc_mean))
# Carte des clusters
df_results <- df_meta %>% mutate(cluster = as.factor(clusters))
ggplot(df_results, aes(x = lon, y = lat)) +
geom_polygon(data = world, aes(group = group), fill = "grey") +
geom_point(aes(color = cluster), shape = 1, alpha = 0.8) +
scale_color_manual(values = c("#a6d854", "#fc8d62", "#756bb1")) +
coord_quickmap() +
theme_minimal()Transformation Hellinger : Essentielle pour les données de composition. Elle normalise les abondances et donne du poids aux espèces rares.
ACP puis clustering : La réduction de dimension avant le clustering améliore la robustesse et élimine le bruit.
Méthode de Ward : Produit des clusters compacts et bien séparés, idéale pour identifier des groupes écologiques distincts.
Ce que j’ai appris
Cette analyse m’a enseigné plusieurs choses sur l’application de la data science à l’océanographie.
Le big data ne suffit pas. Les 6,8 millions d’images n’auraient aucune valeur sans l’expertise océanographique pour les interpréter correctement. C’est la combinaison des deux qui crée de la connaissance.
L’apprentissage non supervisé révèle l’inconnu. Je ne cherchais pas trois communautés spécifiques, c’est le clustering qui les a révélées. Quand on explore des données complexes, ces méthodes sont puissantes pour découvrir des structures cachées.
La validation par le domaine d’expertise est essentielle. Les trois communautés correspondent à des biomes océaniques connus. Sans cette cohérence écologique, les clusters n’auraient été que des artefacts statistiques sans signification.
Enfin, le combo ACP + clustering est un classique pour explorer des données complexes. Il fonctionne particulièrement bien quand vous avez beaucoup de variables corrélées et que vous cherchez des groupes naturels dans vos données.
Références
Pour aller plus loin
Cette analyse fait partie de ma thèse de doctorat sur l’application de l’intelligence artificielle à l’écologie planctonique. Si vous voulez maîtriser les analyses multivariées (ACP, clustering…) et le machine learning avec R, découvrez mes formations.